

1.RNA-seq解析(遺伝子発現解析)のサービス概要
| 価格(税別) | 真核生物(S. cerevisiaeを除く) | 40,000円/サンプル |
|---|---|---|
| Saccharomyces cerevisiae | 60,000円/サンプル 内訳)RNA-seq解析40,000円+rRNA除去(S. cerevisiae用)20,000円 |
|
| 原核生物 | 50,000円/サンプル 内訳)RNA-seq解析40,000円+rRNA除去(原核生物用)10,000円 |
|
| ※S. cerevisiaeと原核生物はオリゴdTでmRNAを精製ができないため、上記の通りrRNA除去が必要となります。 ※8サンプル以上で5%引き、24サンプル以上で10%引き |
||
| データ量 | 4,000万リード/6 Gb | |
| 納期 | 25営業日 ※データ解析不要(生データ納品)の場合は20営業日 | |
| 作業内容 | ・送付サンプル(トータルRNA)の品質確認(注1) ・【真核生物(S. cerevisiaeを除く)】オリゴdTでmRNAを精製 【Saccharomyces cerevisiae・原核生物】各対象生物用のキットを用いてrRNA除去 ・ランダムプライマーでcDNA合成後、シーケンスライブラリー作製 ・シーケンシング解析(DNBSEQ 150 bpペアエンド解析) ・データ解析(注2) Bowtie2/STAR(注3)ソフトウェアを用いて参照配列にマッピングいたします。その後、featureCountを用いてカウントを行った後、TPM正規化を行います。 ※cufflinksを用いた解析も対応可能です。 ※ヒト・マウス・ラット・イヌ・ニワトリ・アナウサギの場合は、リードカウントと正規化の代わりに、 融合遺伝子の検出を行うことも可能です。リードカウントと正規化も行う場合は、10,000円(税別)かかります。 |
|
| 納品物 | 以下のデータをDVDやUSB等の記録媒体に保存して納品します。 ・報告書 ・シーケンス生データ(fastq形式) ・マッピングとカウント結果(エクセルファイル) ・参照配列(fasta形式)と遺伝子アノテーション(gff形式) ・DDBJのデータベース登録に必要なデータ ※データ量が多い場合はHDD納品となり、HDD代として別途20,000円(税別)がかかります。お手持ちのHDDを送付いただき、そちらに保存して納品することも可能です。 |
|
| ご提供サンプル | トータルRNA 1 μg以上(>50ng/ul、>20ul) ※ドライアイスを同梱し、冷凍便を使用して送付ください。 ※RNA濃度が基準値未満の場合は、「微量RNA対応」のオプションをご覧ください。 |
|
| ご依頼の流れ | まずはお見積りをご依頼ください。お見積り依頼フォームをご用意しています。 | |
(注1)rRNAの分解が進んでおり、シーケンシング解析を行ってもデータが得られないと判断した場合は、中止させていただきます(この場合のご請求は行いません)。
(注2)参照配列がない生物種は、De novo transcriptome assembly 解析(オプション/以下参照)を行って発現遺伝子カタログを作成し、参照配列の代わりとしてマッピングに用います。De novo transcriptome assembly 解析を実施しない場合は、生データでの納品となります。
(注3)2024年5月にhisat2からSTARに変更しました。
2.オプション
2-1.オプション(RNA抽出・rRNA除去・微量RNA対応)
| 項目 | 備考 | 価格(税別) | 追加 納期 |
|---|---|---|---|
| RNA抽出(土壌・汚泥以外) | QIAGEN社のRNeasy等を使用して、トータルRNAを抽出します。 <ご提供サンプル量の目安> 細菌:細胞数 1×10^9程度、湿重量 20 mg程度 酵母:細胞数 5×10^7程度、湿重量 25 mg程度 植物(葉):湿重量 50 mg程度、直径1.5cm 動物細胞:細胞数 1×10^7程度、湿重量 20 mg程度 動物組織:湿重量 20 mg程度 <サンプルの保管温度と送付方法> ◇ RNAlaterなどの核酸保存液に浸す場合 採取後すぐにサンプルに核酸保存液を添加します。 一晩は冷蔵保管(4℃)し、翌日に冷凍(-20℃)保管にします。 送付時は、冷凍便を利用し、ドライアイスは同梱せずに送付してください (RNAlater は-20℃以下で凍結してしまうため)。 ◇ 核酸保存液を添加しない場合 採取後すぐにサンプルを-80℃保管します。 送付時は、冷凍便を利用し、必ずドライアイスを同梱してください。 |
20,000円/サンプル | 5営業日 |
| RNA抽出(土壌・汚泥) | RNeasy Powersoil TotalRNA kitを使用して、トータルRNAを抽出したのち、DNase処理を行います。 <サンプル量の目安>土壌:湿重量 1~2 g <サンプルの保管温度と送付方法>上記と同じ |
24,000円/サンプル | 7営業日 |
| rRNA除去(原核生物対象) | Vazyme社のRibo-off rRNA Deletion Kit等を使用して、rRNAを除去します。 | 10,000円/サンプル | なし |
| rRNA除去(S. cerevisiae対象) | siTOOLs Biotech社のriboPOOL等を使用して、rRNAを除去します。 | 20,000円/サンプル | なし |
| 微量RNA対応(真核生物のみ可) | 微量RNA(>1 ng/ul、>20 ul)に対して、ライブラリー調製を行います。劣化したRNAではライブラリー調製ができません。 また、微量RNA対応は通常のライブラリー調製方法と異なるため、複数サンプルをご依頼いただく際は、どちらかの調製方法で統一することを強く推奨します(統一しないと、サンプル間の発現量の比較ができなくなります)。 |
20,000円/サンプル | 5営業日 |
2-2.オプション(データ解析)
★2024.09 目的別にパッケージ化しました★
| 項目 | 備考 | 価格(税別) | 追加 納期 |
|---|---|---|---|
| 発現プロファイルの類似性 | 発現比較解析前に各サンプルの遺伝子プロファイルの類似性をいくつかの手法で可視化し、反復内や反復間で遺伝子発現の差を確認します。 (主成分分析、相関分析、階層的クラスタリング) |
30,000円 ※3サンプル以上で解析可 |
なし |
| 発現比較解析 | 統計解析によって発現量が有意に異なる遺伝子群を特定した後、それをMA plotやVolcano plot、ヒートマップで描写します。 | 1比較目30,000円 ※追加1比較ごと+10,000円 |
なし |
| ┗ エンリッチメント解析 | 発現変動した遺伝子群が、どのような機能をもつのか体系的に分類し、有意差検定を行います。また、KEGGの代謝マップ上に可視化するための情報を納品します。 (Gene Ontology(GO)解析、パスウェイ解析、KEGG pathway classification chart) |
1比較目50,000円 ※追加1比較ごと+10,000円 |
なし |
| De novo transcriptome assembly | 参照配列がない非モデル生物の遺伝子カタログ配列を、Trinityで構築します。 | 100,000円/生物種 | なし |
3.具体例
標準解析
遺伝子発現行列データをエクセルデータで納品します。>>詳しいデータをご覧いただけます
サンプル毎にどの遺伝子に何リードマッピングされたかを示しています。また、TPM正規化後の数値も示しています。
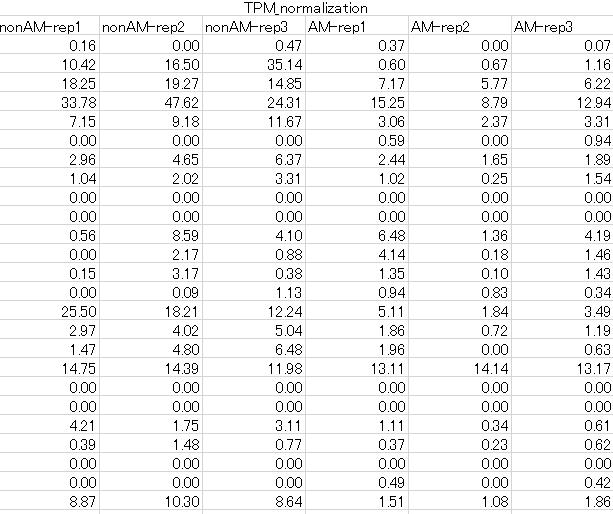
発現プロファイルの類似性(オプション)
主成分分析
主成分分析の結果をpng形式で納品します。
相関図
相関図をpng形式で納品します。
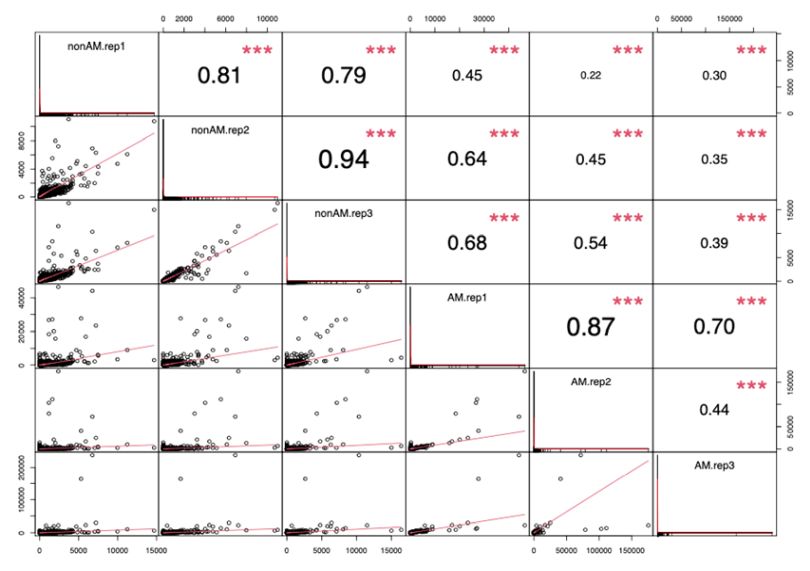
===図の説明===
右上の部分の各数字がpearson(ピアソン)の相関関数です。
数字右上の * はp-valueです。* <0.05, ** <0.01, *** <0.001
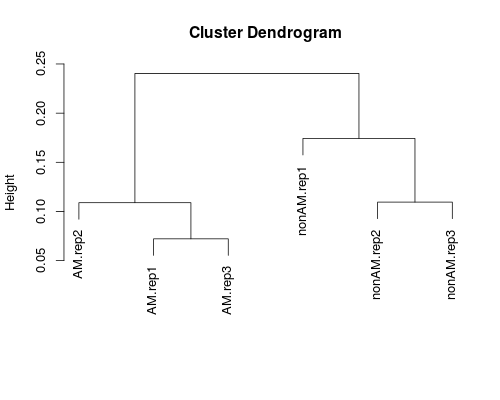
階層的クラスタリング
クラスタリングの結果をpng形式で納品します。
発現比較解析(オプション)
1.遺伝子発現比較解析の結果をエクセルデータで納品します。>>詳しいデータをご覧いただけます
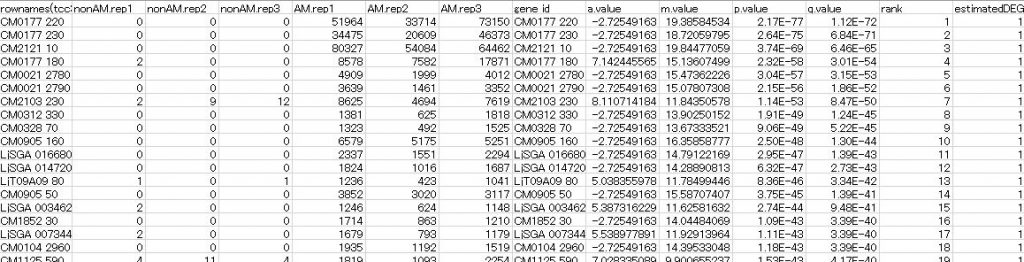
===データの見方===
-sample_name: 正規化前のリードカウント
– a.value: log2(平均発現量)
– m.value: log2(発現比)
– p.value: p値
– q.value: q値 (false discovery rate(FDR))
– rank: p値が低い順にランク付けされています。
– estmatedDEG: 「1」は発現変動遺伝子(q<0.05)、「0」は発現変動遺伝子ではないこと(q>0.05)を示します。
2.MAプロットの図をpng形式で納品します。
横軸がlog2(平均発現量(A))、縦軸がlog2(発現比(M))を示しています。
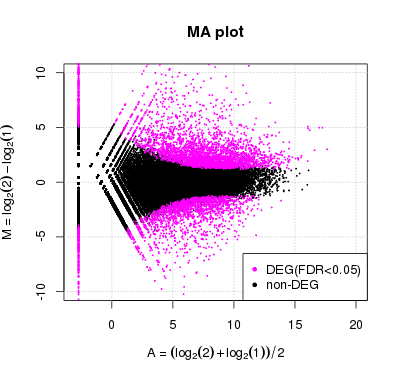
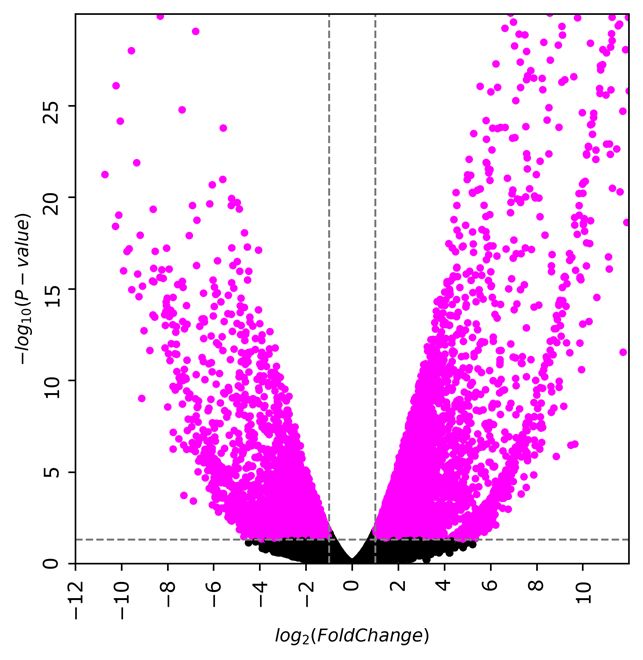
Volcanoプロット
Volcano プロットをpng形式で納品します。
Volcano プロット は、MAプロットの親戚の図です。volcano plotは横軸がlog2(発現比)、縦軸がp値を示しています。
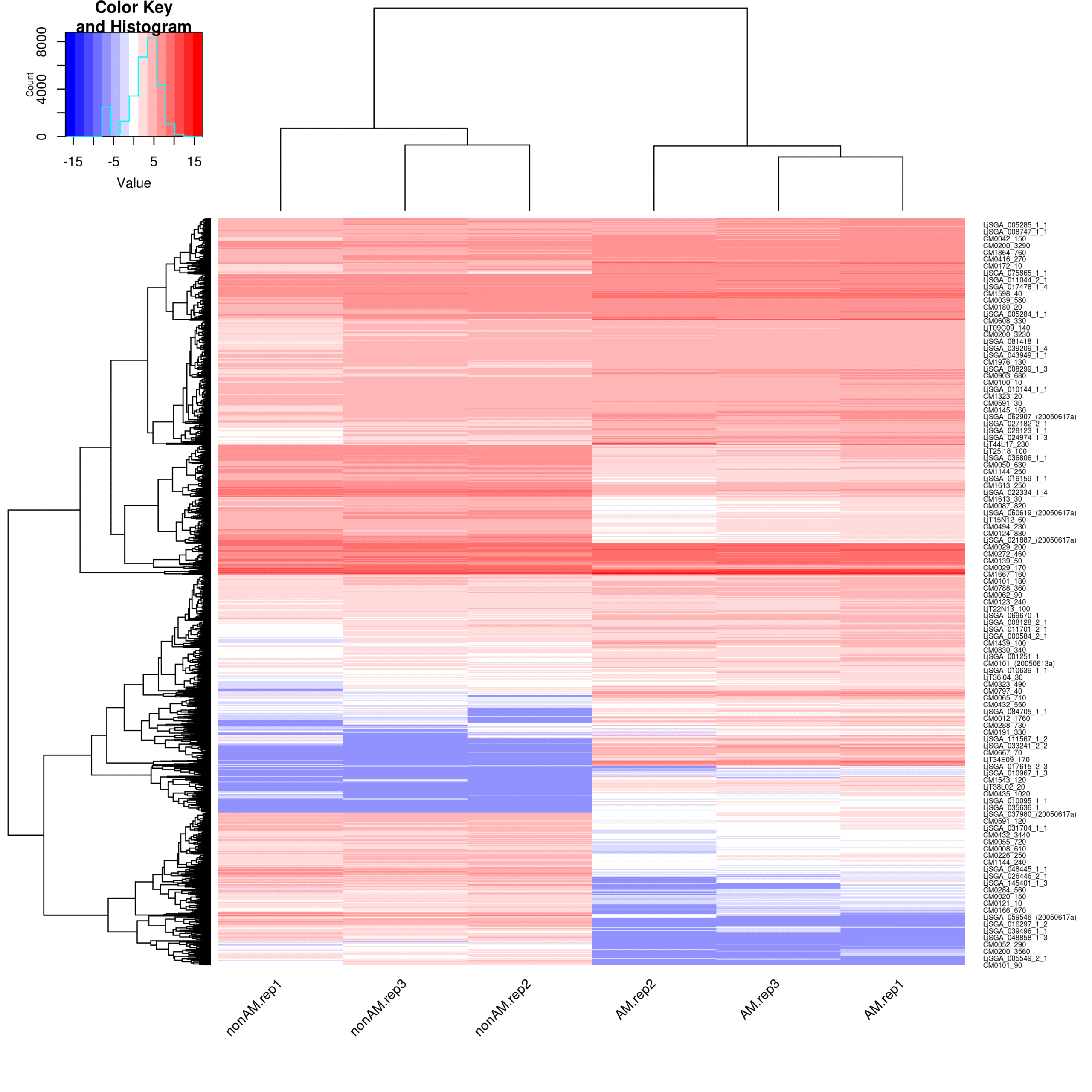
ヒートマップ
ヒートマップ図をpng形式で納品します。
エンリッチメント解析
パスウェイ解析
パスウェイ解析の結果をテキストデータで納品します。エクセルで開くことができます。>>詳しいデータをご覧いただけます

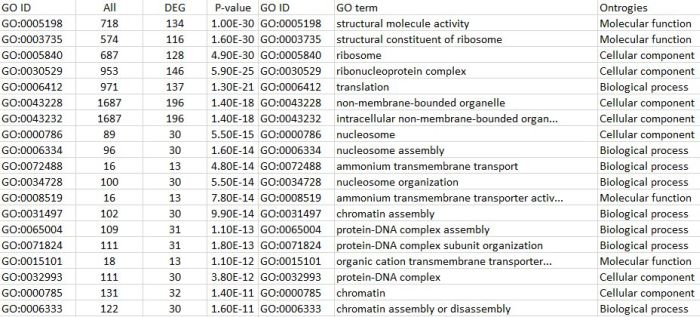
Gene Ontology(GO)解析
Gene Ontology(GO)解析の結果をエクセルデータで納品します。
4.よくある質問
Q1.推奨のRNA抽出キットは?
特にございません。弊社ではQIAGEN社のRNeasyシリーズを使用することが多いです。
Q2.DNase処理は必要?
ゲノムDNAの混入は望ましくないので、DNase処理をお勧めします。
Q3.送付の際、バッファーは何でもよい?
滅菌水のほか、Tris や TE 、抽出キットの溶出バッファーでも構いません。但し、塩濃度が高いバッファーの場合、QCの電気泳動がうまくいかない可能性がありますので、避けてください。
Q4.RIN値の目安は?
バイオアナライザーのRIN値で7以上が目安です。送付前にお客様側でも品質確認される際は、参考にしてください。
Q5.送付時の温度帯は?
●total RNA送付の場合
冷凍便を利用し、必ずドライアイスを同梱してください。
●生サンプル送付(弊社にRNA抽出もご依頼)の場合
◇RNAlaterなどの核酸保存液に浸しているサンプル
冷凍便を利用し、ドライアイスは同梱せずに送付してください(核酸保存液は-20℃以下で凍結してしまうため)。
◇-80℃で保存していたサンプル
冷凍便を利用し、必ずドライアイスを同梱してください。
Q6.ウイルスの解析は可能?
以下の通り、ゲノムの形状により解析できるものとできないものがあります。
一本鎖DNA(ssDNA):×
二本鎖DNA(dsDNA):〇
一本鎖RNA(ssRNA):〇
二本鎖RNA(dsRNA):〇 ※環状化dsRNAは×
また、①宿主由来のrRNAの混入があるか、②対象ウイルスのRNAにpolyAがあるかによって、ライブラリー調製方法が変わりますので、お見積り依頼時に①②の情報をお知らせください。
Q7.オプションのデータ解析は必要?
必要かどうかは目的によります。
オプションを付けない標準のRNA-seq解析でも遺伝子の発現量やざっくりとした遺伝子発現の違いは明らかにできます。
それが統計的に有意かどうか解析する場合に「発現比較解析」が必要になります。
「発現比較解析」は発現量に有意差のある遺伝子(発現変動遺伝子)をリストアップするだけですので、同定された発現変動遺伝子の代謝系における役割や機能的グループを明らかにしたい場合は、「エンリッチメント解析(パスウェイ解析やGO解析)」が必要になります。「パスウェイ解析」では発現変動遺伝子が代謝系のどの部分で変動しているのかが分かり、「GO解析」では発現変動遺伝子がどのような機能をもったグループが多いかが分かります(出現頻度の解析)。
Q8.発現比較解析などの比較数って?

3群間比較(A群 VS B群 VS C群)も可能ですが、発現比較の発現平均値(A値)と発現比(M値)が算出されないため、MAプロットとVolcanoの図の納品はなく、エクセルデータのみの納品となります。
Q9.納品後にデータ解析を追加で依頼できる?
ご依頼いただけます。データの保管期間は納品後3か月ですので、それ以降はデータ送付をお願いする場合がございます。
Q10.DNBSEQの特徴は?
なんといってもコストパフォーマンスがよいことです。低価格で高品質のショートリードが取得できます。
Q11.DNBSEQの出力データはどんな形式?
出力はfastqファイルで、Q値は最新のイルミナ社製シーケンサーと同様にsanger形式です。
Q12.DNBSEQの生データのクオリティは?
DNBSEQ-G400(MGI社)とHiSeq(イルミナ社)を用いてシーケンシング解析を行い、取得したリードのQ20およびQ30を比較したところ、2機種のそれらは同等でした。
5.ちらしPDF
6.お問合せ・サンプル送付先
お見積り・お問合せ
お見積りフォーム、お問合せフォーム、メール(dna@gikenbio.com)、または お電話(042-780-8333)でお気軽にご相談ください。
サンプル送付先
株式会社生物技研
〒252-0154 神奈川県相模原市緑区長竹657 / TEL 042-780-8333
※平日着でお送りください。発送は元払いでお願いしています。
<添付書類>
・依頼書→dna@gikenbio.comへ送信ください。あわせてサンプル送付時に印刷したものを同封ください。





